|
|
 |
Tutorial-2:
Das Grippemedikament TAMIFLU wirkt als Neuraminidasehemmer
made with
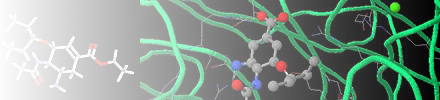
| Neuraminidase mit eingelagertem
Tamiflu
(aus:
rcsb.org)
- |
In Jmol lassen
sich Teilstrukturen durch eine sog."Halomarkierung" hervorheben. Hier wird
der Neuraminidasehemmstoff, der als Grippemedikament entwickelt wurde
. Bei näherer Betrachtung sieht man, dass sich das Wirkstoffmolekül
in einer "Tasche" nahe der Oberfläche des globulären Enzyms befindet.
Zur besseren Sichtbarkeit wird das kleine Molekül in
dargestellt. Eine atomrichtige Darstellung (CPK-Mode) wird am Ende angeboten.
Die "Tasche" im Enzym Neuraminidase wird noch besser sichtbar, wenn man
das "Rückgrat" des Proteins mit einer
beschreibt und die einzelnen Atome der Aminosäureseitenketten durch
ein "Drahtgitter" ersetzt. Anschließend lassen sich nur die Seitenketten
von
sowie von
darstellen. Diese Aminosäurereste kommen gehäuft in der Umgebung der "Tasche" vor und tragen zur Bindung eines Neuraminidasehemmstoffs bei. wechselwirken mit der , mit der von TAMIFLU. Durch Drehung des Moleküls mit der Maus lässt sich die Bindungstasche gut erkunden. Zur atomrichtigen Darstellung des Hemmstoffs wird der und eine aktiviert. Ein Vergleich mit der folgenden Darstellung (Neuraminidase mit Sialinsäure) zeigt, dass der Hemmstoff im aktiven Zentrum des Enzyms angreift und damit eine kompetitive Hemmung bewirkt.
|
| Neuraminidase mit eingelagerter
Sialinsäure
(aus:rcsb.org)
|
Die Sialinsäure
(N-Acetylneuraminsäure) ist das eigentliche Substrat der viralen Neuraminidase.
Sie ist endständiger Teil von Rezeptoren der Wirtszellen, die von den
Hämagglutinin-Oberflächenproteinen der Viren erkannt werden.
Mit Hilfe des Kontextmenues des JSmol-Fensters (Rechtsklick auf die Abbildung) kann man versuchen, eine Ansicht zu erstellen, die der der obigen Tamiflu-Einlagerung entspricht (ausprobieren!). Zunächst sollte man die Ansicht und die Größe vergleichbar der Neuraminidase-Tamiflu-Darstellung einstellen. Das Calcium-Ion (grün) kann zur Orientierung dienen. Anschließend wählt man das Substrat mittels AUSWAHL-- HETERO--NACH HETAM--SIA-O-SIALIC ACID aus und markiert es mit einem Halo. |
| Die hydrolytische Abspaltung der Sialinsäure
(Strukturdatei aus:rcsb.org) Hier kommen die benachbarten Aminosäurereste
des Enzyms ins Spiel. Diese
können mit der positiven Ladung am Ringsauerstoffatom wechselwirken
und sie so stabilisieren. |
Um eine Vorstellung zu entwickeln, wie die hydrolytische Abspaltung der endständigen Sialinsäure durch das Enzym bewerkstelligt wird und eine mechanistische Verbindung zur Hydrolyse eines Vollacetals im Rahmen der Konzepte der organischen Chemie zu schaffen, muss man die strukturelle Umgebung im aktiven Zentrum genauer betrachten. Dazu werden zunächst vier wichtige Aminosäurereste dargestellt. Die von J. N. Varghese et.al. 1992 in der PDB-Datenbank veröffentlichte Struktur, die auf kristallografischen Daten beruht, enthält aus experimentellen Gründen nicht das wirkliche Substrat der Neuraminidase (ein Polysaccharid mit Sialinsäure als endständiger Gruppe), sondern nur die Sialinsäure. Wenn man das enzymatische Geschehen verstehen will, muss man beachten, dass die OH-Gruppe an C2 diejenige ist, die mit einem weiteren Saccharidbaustein im Sinne einer glycosidischen Bindung verknüpft ist. Zur besseren Orientierung in der Jmol-Darstellung wird das O-Atom an C2, das die glykosidische Bindung zu dem nicht dargestellten Oligosaccharidrest des Rezeptors bildet, vergrößert dargestellt und die Ansicht . (Dieses O-Atom "vertritt" in der Visualisierung den nicht dargestellten Rest des Rezeptors). Bei der Abspaltung der Sialinsäure vom Rest des Polysaccharids muss die Bindung zwischen C2 und O2 hydrolytisch gespalten werden. Die entscheidenden Wechselwirkungen, die zu einer enzymatisch beschleunigten Hydrolyse dieser Bindung führen, finden in der Region zwischen den Aminosäureresten und der Region zwischen den Aminosäureresten statt. Es gibt gute Hinweise dafür, dass der saure Aminosäurerest Asp151 unter Mitwirkung von Arg 152 ein Wassermolekül (in der Jmol Struktur nicht dargestellt) bindet, welches ein Proton auf das , das die glycosidische Bindung ausbildet, überträgt (hier durch den Pfeil dargestellt). Dieser Schritt entspricht der Protonierung eines Sauerstoffatoms eines Vollacetals als erstem Schritt der hydrolytischen Abspaltung des Alkohols unter Ausbildung eines Carbeniumions. Nach der Bindung zwischen dem C2-Atom und dem O2-Atom und Freisetzung des Polysaccharidrests des Rezeptors trägt das C2-Atom formal eine positive Ladung. Die Ausbildung eines Carbeniumions mit einer positiven Ladung an C2 wird stabilisiert durch , die durch ein freies Elektronenpaar des Ringsauerstoffatoms zur Verfügung gestellt wird. Beim eines freien Elektronenpaars und Ausbildung einer formalen Doppelbindung zwischen C2 und dem Ringsauerstoff verlagert sich die positive Ladung an das Sauerstoffatom. In diesem Zustand kommt es entscheidend darauf an, dass diese positive Ladung durch negative Ladungsdichte in der Umgebung ausgeglichen werden kann. Diese Möglichkeit erst beschleunigt den geschwindigeitsbestimmenden Schritt der Hydrolyse (weiter linke Spalte!) |